크로마틴 접근성 분석을 위한 ATAC-Seq
ATAC-Seq(시퀀싱을 이용한 전위효소 접근성 크로마틴 Assay)
고처리량 역량으로 유전체 전체에 걸친 액세스 가능한 크로마틴을 프로파일링하기 위한 빠르고 민감한 방법
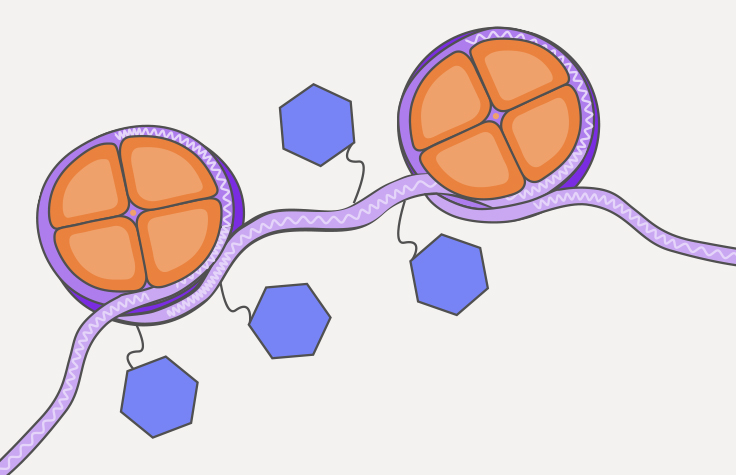
ATAC-Seq이란?
ATAC-Seq(assay for transposase-accessible chromatin with sequencing)은 유전체 전체의 크로마틴 접근성 평가를 위한 주요 방법입니다. ATAC-Seq을 사용하면 열린 크로마틴 영역을 시퀀싱해 크로마틴 포장을 비롯한 여타 요인이 유전자 표현형에 어떤 영향을 미치는지 알아낼 수 있습니다.
ATAC-Seq은 조절 요소에 대한 사전 지식이 필요 없는 강력한 후성유전학적 발견 도구입니다. 복합 질환, 배아 발생, T세포 활성화 및 암에서 크로마틴 접근성, 전사 인자 결합 및 유전자 조절을 더 잘 이해하는 데 사용되어 왔습니다.1,2 ATAC-Seq은 벌크 세포 집단 또는 단일세포에서 고해상도로 수행할 수 있습니다.
ATAC-Seq의 작동 방식
ATAC-Seq에서는 genomic DNA가 활동 과잉 전위효소 Tn5에 노출됩니다. Tn5는 DNA를 동시에 절편화하고 열린 크로마틴 부위에 우선적으로 삽입(insert)하며 시퀀싱 프라이머를 추가합니다(태그멘테이션으로 알려진 과정). 시퀀싱된 DNA를 통해 열린 크로마틴을 확인하고 이러한 데이터 분석은 유전자 조절에 대한 통찰을 제공할 수 있습니다.
또한 ATAC-Seq은 RNA 시퀀싱과 같은 다른 방법과 결합하여 유전자 발현 연구에 대한 멀티오믹스 접근 방식을 사용할 수 있습니다.3,4 후속 실험에는 종종 ChIP-Seq, Methyl-Seq 또는 Hi-C-Seq이 포함되어 후성유전학적 조절 형태를 더욱 자세하게 규명합니다.
ATAC-Seq를 단일세포에 적응시키는 방법
William Greenleaf 박사가 ATAC-seq의 개발 및 단일세포에 대한 적응에 대해 논의합니다. 또한, 유전체학 및 후성유전학 전문가인 Bing Ren 박사와 Sebastian Preissl 박사는 복합 색인화를 사용하여 단일세포 ATAC-Seq에 대해 논의합니다.
웨비나 시청하기
주요 ATAC-Seq 애플리케이션
ATAC-Seq을 사용한 크로마틴 접근성 분석은 유전체의 조절 환경에 대한 귀중한 통찰을 제공할 수 있습니다. 주요 애플리케이션에는 다음이 포함됩니다.
- 뉴클레오솜 매핑
- 전사 인자 결합 분석
- 새로운 인핸서 식별
- 질병 관련 조절 메커니즘 탐색
- 세포 타입별 조절 분석
- 진화 연구
- 비교 후성유전학
- 생체표지자 발견
ATAC-Seq를 사용한 출판물
T세포에서 유전자 조절
과학자들이 ATAC-Seq를 사용하여 유전자 발현을 조절하기 위해 크로마틴 상태에 영향을 미치는 조절인자를 식별하는 방법을 읽어보세요.
ATAC-Seq를 사용하는 개선된 메타유전체학적 어셈블리
연구자들이 ATAC-Seq의 실행 가능성과 이점을 입증하여 세균 유전체에 대한 연구를 지원하는 방법을 알아보세요.
암에서 유전체 구조 특성 확인
과학자들이 The Cancer Genome Atlas의 15가지 원발성 인간 암 유형을 사용하여 암의 유전체 토폴로지를 더 잘 이해하기 위해 ATAC-Seq를 활용하는 방법을 알아보세요.

차세대 시퀀싱을 통한 유전자 조절 및 전사체학 연구 가속화
RNA-Seq이 다양한 분야에서 유전자 조절 및 전사체 연구를 어떻게 발전시키고 있으며, 유전자 조절 연구를 통해서 어떻게 상호 보완적인 정보를 얻을 수 있는지 확인해 보세요.
eBook 다운로드ATAC-Seq에 대한 커버리지 권장사항
ATAC-Seq에 필요한 최소 시퀀싱 커버리지는 연구 목적에 따라 다릅니다. 이 표는 일반적인 애플리케이션에 대한 몇 가지 가이드라인을 제공합니다.
ATAC-Seq용 페어드 엔드 리드(paired-end·reads) 사용을 권장합니다. 싱글 리드 시퀀싱과 비교할 때 페어드 엔드 리드는 다음을 제공합니다.
- 더 높은 고유 정렬 비율
- PCR 중복 제거 개선
- 접근 가능한 시퀀스에 대한 더욱 완전한 정보
- 리드를 뉴클레오솜 프리, 단일 뉴클레오솜 또는 디뉴클레오솜으로 분류하는 더 높은 정밀도
| 연구 목표 | 권장 뎁스(depth) |
|---|---|
| 인간 샘플의 열린 크로마틴 차이 식별 | ≥ 50M 페어드 엔드 리드 |
| 유전자 조절 네트워크 구성을 위한 전사 인자 풋 프린팅 | > 200M 페어드 엔드 리드 |
| 단일세포 분석 | 핵/세포당 25~50 K 페어드 엔드 리드 |
실험 요건이 다양할 수 있으므로 과학 문헌을 참조하여 프로젝트에 적합한 커버리지 수준을 결정하는 것이 좋습니다.
ATAC-Seq을 사용하면 이전에는 전체 유전체 수준에서 관찰할 수 없었던 복잡하거나 희귀한 조직의 후성유전학적 변이성과 세포 집단의 후생유전학적 환경에 대해 질문할 수 있습니다.
추가 리소스
전사체 및 후성유전체 프로파일링
이 기술 노트는 10x Genomics Chromium Single Cell Multiome ATAC + Gene Expressions을 사용하여 단일 세포로부터 전사체 및 후성유전체의 동시 프로파일링을 기술합니다.
The regulatory landscape of A. thaliana
애기장대의 단일세포 ATAC-Seq에 대한 이 웨비나에 참여해 보세요. 발표자는 식물 발달을 관장하는 조절 네트워크를 추론하기 위한 분석 프레임워크에 대해 논의합니다.
단일세포 애플리케이션에 ATAC-Seq 최적화
이 웨비나에서 업계 전문가들은 scRNA-Seq, sNuc-Seq, ATAC-Seq 및 기타 assay의 샘플 준비 기법과 모범 사례에 대한 통찰력을 제공합니다.
암 후성유전학
ATAC-Seq를 사용하여 암의 후성유전학적 특징을 탐색하는 방법과 함께 시작하는 데 도움이 되는 가이드, 제품 및 기타 리소스를 살펴보세요.
ATAC-Seq에 대한 FAQ
참고 문헌
- Cusanovich DA, Reddington JP, Garfield DA, et al. The cis-regulatory dynamics of embryonic development at single-cell resolution. Nature. 2018; 555:538-542. 13.
- Gate RE, Cheng CS, Aiden AP, et al. Genetic determinants of co-accessible chromatin regions in activated T-cells across humans. Nat Genet. 2018; 50:1140-1150.
- Luo L, Gribskov M, Wang S. Bibliometric review of ATAC-Seq and its application in gene expression. Briefings Bioinform. 2025;23:1-13. doi: 10.1093/bib/bbac061.
- Cao J, Cusanovich DA, Ramani V, et al. Joint profiling of chromatin accessibility and gene expression in thousands of single cells. Science. 2018;361:1380-1385.