
DNA 메틸화 eBook
DNA 메틸화를 이해하는 것은 인간 질환 및 기타 표현형의 연구에 중요합니다. 이 eBook은 DNA 메틸화를 분석하고 시험 설계, 샘플 유형, 데이터 분석을 포함한 전장 후성유전체 연관성 연구를 수행하기 위한 고려사항을 제공합니다.
DNA 메틸화 eBook
귀하의 이메일 주소는 결코 제3자와 공유되지 않습니다.
DNA 메틸화 시퀀싱을 위해 NGS 기술을 이용한 바이설파이트 시퀀싱
바이설파이트 시퀀싱(BS-Seq) 또는 전장 유전체 바이설파이트 시퀀싱(WGBS)은 유전체 DNA에서 메틸화 시토신을 검출하기 위한 잘 확립된 프로토콜입니다. 이 방법에서는 유전체 DNA를 바이설파이트나트륨으로 처리한 다음 시퀀싱하여, 유전체에서 메틸화 시토신의 단일 염기 해상도를 제공합니다. 바이설파이트나트륨으로 DNA를 처리한 후, 비메틸화 시토신은 우라실로 탈아미노화되고, 시퀀싱 후 티민으로 전환됩니다. 동시에, 메틸화 시토신은 탈아미노화에 저항하며 시토신으로 판독됩니다. 처리된 염기서열과 처리되지 않은 염기서열을 비교하여 메틸화 시토신의 위치를 결정합니다.1,2


DNA 메틸화 eBook
DNA 메틸화를 이해하는 것은 인간 질환 및 기타 표현형의 연구에 중요합니다. 이 eBook은 DNA 메틸화를 분석하고 시험 설계, 샘플 유형, 데이터 분석을 포함한 전장 후성유전체 연관성 연구를 수행하기 위한 고려사항을 제공합니다.
귀하의 이메일 주소는 결코 제3자와 공유되지 않습니다.

유전자 발현 및 조절 eBook
유전자 조절은 mRNA와 비코딩 RNA 전사물을 포함한 유전자 산물의 시간적, 공간적 발현을 조절하는 생물학적 과정입니다. BS-Seq 및 NGS 기술을 포함한 현대적인 전사체학 및 후성유전학 방법을 사용하여 어떻게 발견을 하고 이 생물학적 과정을 보다 잘 이해할 수 있는지 알아보십시오.
귀하의 이메일 주소는 결코 제3자와 공유되지 않습니다.

Power of Multiomics eBook
DNA는 분자 정보를 유전적 염기와 후생유전학 염기에 저장하여 본질적으로 멀티오믹스을 만듭니다. RNA-Seq의 전사체 정보를 메틸화 시퀀싱 데이터와 결합하면 세포 분화, 메커니즘 및 거동을 더 잘 이해할 수 있습니다. 멀티오믹스 접근 방식의 힘을 자세히 알아보고 활용하십시오.
귀하의 이메일 주소는 결코 제3자와 공유되지 않습니다.
축소 표현 바이설파이트 시퀀싱(RRBS-Seq) 또는 단일세포 축소 표현 바이설파이트 시퀀싱(scRRBS)은 서열 특이적 절편화를 생성하기 위해 유전체 DNA에 하나 또는 여러 제한 효소를 사용하여 하는 프로토콜입니다. 그런 다음, 단편화된 유전체 DNA를 바이설파이트로 처리하고 시퀀싱합니다. RRBS-Seq는 특정 관심 영역을 연구하기 위해 선택하는 방법입니다. 이 방법은 프로모터 및 반복 영역과 같이 메틸화가 높은 경우에 특히 효과적입니다.3,4
산화 바이설파이트 시퀀싱(oxBS-Seq)은 5mC와 5hmC를 구분합니다. oxBS 사용 시, 5hmC는 산화제에 의해 5-포르밀시토신(5fC)으로 산화되는 반면, 5mC는 변하지 않습니다. 산화된 5hmC를 바이설파이트나트륨으로 처리하면 우라실로 탈아민화되며, 이는 시퀀싱 시 티민으로 판독됩니다. oxBS-처리된 DNA를 딥 시퀀싱하고 처리된 DNA와 미처리 DNA의 염기서열을 비교하면 염기 해상도에서 5mC 위치를 식별할 수 있습니다.5,6
태그멘테이션 기반 전장 유전체 바이설파이트 시퀀싱(T-WGBS)은 Tn5 전위효소와 중바이설파이트 전환을 사용하여 5mC를 연구하는 프로토콜입니다. 표준 WGBS 방법과 비교하여, T-WGBS는 최소 DNA(약 20 ng)로 물질을 시퀀싱할 수 있습니다. 또한, Tn5 전위효소는 단 한 단계로 DNA 절편화 및 시퀀싱 어댑터 부착의 이점을 제공합니다. 그런 다음 DNA를 바이설파이트나트륨으로 처리하고 PCR 증폭한 후 시퀀싱합니다.7
단일세포 바이설파이트 시퀀싱(scBS-Seq)은 단일세포로부터 게놈 DNA 내 메틸화 시토신을 검출하도록 수정된 잘 확립된 바이설파이트 시퀀싱(BS-Seq) 및 바이설파이트 후 어댑터 태그 부착(PBAT) 프로토콜의 버전입니다. 이 방법에서는 단일세포를 단리한 후, 게놈 DNA를 바이설파이트나트륨으로 처리하여 DNA를 절편화합니다. 그런 다음, 전환된 DNA는 여러 번 무작위 프라이밍을 거치고 시퀀싱을 위해 PCR 증폭됩니다. 딥 시퀀싱은 단일세포에서 메틸화 시토신의 고해상도 단일 뉴클레오티드 해상도를 제공합니다.8
| 방법 | 바이설파이트 시퀀싱의 장점 |
바이설파이트 시퀀싱의 단점 |
|---|---|---|
| BS-Seq |
|
|
| RRBS-Seq / scRRBS |
|
|
| oxBS-Seq |
|
|
| T-WGBS |
|
|
바이설파이트 시퀀싱은 시퀀싱 데이터에서 바이설파이트를 사용하여 비메틸화 C를 T로 전환하는 DNA 메틸화 검출에 일반적으로 사용되는 방법입니다. 그러나 이 방법에는 여러 한계가 있습니다. 유전체 내 CpG 부위의 약 10%는 바이설파이트 전환 후 정렬이 어려울 것으로 추정되며, DNA 분해가 90% 수준에 달할 수 있습니다.9
바이설파이트 시퀀싱의 대안인 Illumina 5-염기 솔루션은 새로운 chemistry 및 분석을 활용하여 유전자 변이 및 메틸화 검출을 동시에 지원합니다. Illumina 5-염기 chemistry는 DNA에 손상을 주지 않고 라이브러리 복잡성을 유지하는 간단한 단일 단계 과정에서 5mC를 T로 직접 변환합니다. Illumina 5-염기 용액은 단일 assay로 비변형 염기(A, T, G, C) 및 5mC를 판독할 수 있습니다.
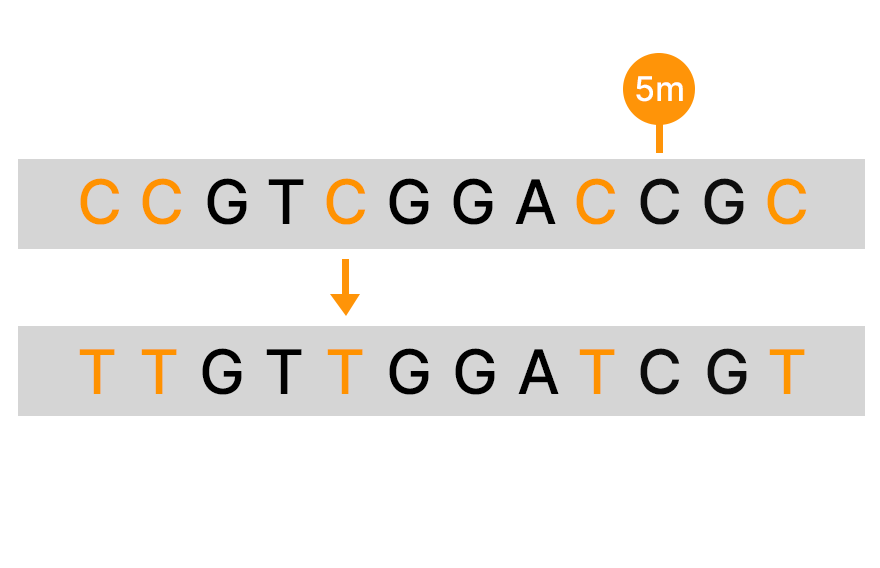
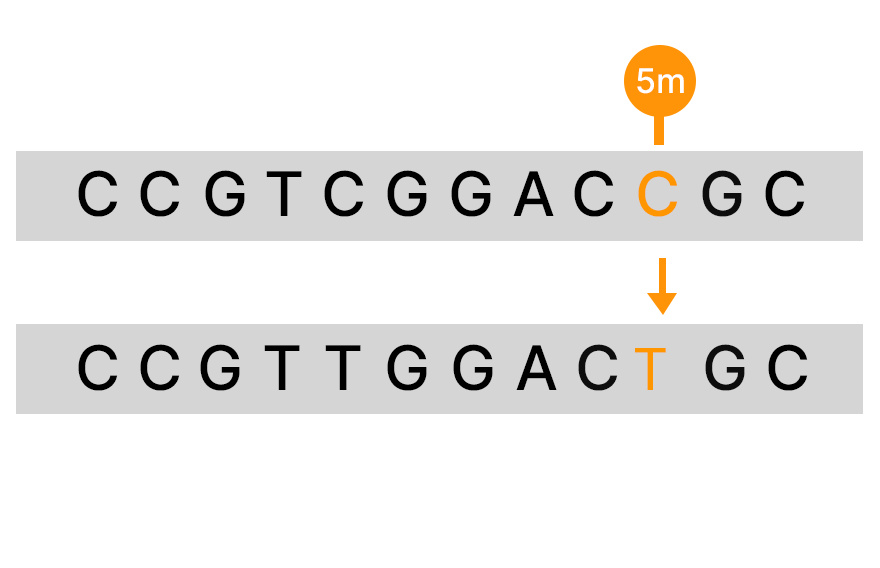
바이설파이트 전환은 바이설파이트나트륨이 선택적으로 비메틸화 시토신을 우라실로 전환시키는 반면, 메틸화 시토신은 전환되지 않은 상태로 유지되는 과정입니다. 바이설파이트 처리는 전장 유전체 DNA 메틸화 프로파일링을 가능하게 하기 위해 NGS와 동시에 흔히 사용됩니다. NGS의 기본 사항에 대해 더 알아보기.
바이설파이트 시퀀싱은 DNA 메틸화를 연구하는 데 일반적으로 사용되는 방법입니다. 시퀀싱 전, 바이설파이트나트륨 처리를 통해 비메틸화 시토신이 우라실로 전환됩니다. 그런 다음 바이설파이트 처리된 샘플과 처리되지 않은 샘플에서 시퀀싱된 DNA를 비교하여 메틸화 및 비메틸화 시토신의 위치를 식별합니다.
바이설파이트나트륨으로 DNA를 처리하면 화학적 탈아미노화에 의해 비메틸화 시토신이 우라실로 전환되는 반면, 메틸화 시토신은 이 과정으로부터 보호됩니다.
DNA 분해, 유전체 복잡성 감소, 정렬 과정에서 추가 복잡성을 포함해 바이설파이트 전환 과정과 후속 시퀀싱에서 발생하는 몇 가지 한계가 있습니다.10
표적 BS-Seq는 바이오마커 발견에 사용하기 위해 연구되었으며, 특정 관심 유전체 절편을 포착하기 위해 비오틴화 RNA 프로브와 같은 구성요소를 사용합니다. 반대로, WGBS는 전체 유전체의 시퀀싱 및 분석으로 귀결됩니.11
NGS 플랫폼에서 DNA 메틸화 연구를 지원하는 당사의 도구와 리소스를 살펴보십시오.
바이설파이트 전환 시퀀싱을 포함한 메틸화 시퀀싱의 장점에 대해 알아보세요.
복잡한 생물학적 메커니즘에 대한 더 나은 인사이트를 얻기 위해 멀티오믹스 분석의 힘을 활용하세요.
참고 문헌
메틸화 연구를 위한 솔루션에 대해 자세히 알아보려면 전문가와 상의하십시오.
귀하의 이메일 주소는 결코 제3자와 공유되지 않습니다.